Kháng kháng sinh – thách thức y tế toàn cầu
Kháng sinh từng là vũ khí tối thượng trong cuộc chiến chống lại vi khuẩn, nhưng thế chủ động của y học đang dần bị đe dọa. Sự gia tăng nhanh chóng của các chủng vi khuẩn mang gen kháng kháng sinh, đặc biệt là những gen chưa từng được ghi nhận trước đó, đang đặt ra bài toán nan giải cho cả điều trị lẫn dự phòng.
Để đối mặt với thách thức này, nhóm các nhà khoa học Việt Nam đến từ Học viện Quân y đã triển khai một dự án nghiên cứu mang tính chiến lược, ứng dụng giải trình tự ADN toàn bộ hệ gen kết hợp với phân tích dữ liệu lớn để truy vết, nhận diện và dự báo xu hướng kháng thuốc của vi khuẩn. Dự án do được kỳ vọng mở ra hướng đi mới trong giám sát và kiểm soát dịch tễ học di truyền.
Bức tranh di truyền: Những xu hướng nguy hiểm đang nổi lên
Phân tích loạt các chủng vi khuẩn được phân lập từ các bệnh phẩm lâm sàng, nhóm các nhà khoa học ghi nhận nhiều dấu hiệu cảnh báo cho thấy vi khuẩn ngày càng trở nên khó kiểm soát hơn. Trung bình mỗi chủng mang hơn 10 gen kháng thuốc [1] – một con số đáng lo ngại, và thậm chí có những chủng đạt tới mức cao hiếm thấy trước đây. Điều đặc biệt đáng chú ý là xu hướng này không đứng yên mà đang gia tăng rõ rệt theo thời gian, từ số lượng gen cho đến mức độ đa dạng và phổ kháng thuốc.
Thêm vào đó, một số gen kháng mạnh như aadA2 (kháng aminoglycoside) và blaNDM-5 (kháng carbapenem) chỉ xuất hiện trong các mẫu được thu thập ở giai đoạn sau mà không được phát hiện trong các mẫu từ giai đoạn trước của nghiên cứu trên, cho thấy những biến thể mới đang âm thầm xuất hiện và lan rộng mà không thể bị phát hiện nếu không có công nghệ giải trình tự hiện đại. Những dữ liệu này là lời cảnh tỉnh rõ ràng rằng kháng thuốc đang âm thầm leo thang và có thể bùng phát bất cứ lúc nào nếu không có các biện pháp giám sát chủ động và chính xác.
Khám phá nổi bật: Một gen kháng mới chưa từng được ghi nhận
Cuộc điều tra bắt đầu từ một chi tiết bất thường. Một chủng vi khuẩn thể hiện khả năng kháng mạnh với Imipenem (kháng sinh carbapenem) – một loại kháng sinh vốn được xem là “tuyến phòng thủ cuối cùng” trong điều trị nhiễm khuẩn nặng. Thế nhưng, khi phân tích toàn bộ hệ gen của chủng này, nhóm nghiên cứu không tìm thấy bất kỳ gen kháng carbapenem nào đã được ghi nhận trong các cơ sở dữ liệu quốc tế. Khoảng trống đó không thể giải thích bằng các giả thuyết thông thường.
Không dừng lại ở việc ghi nhận sự vắng mặt, nhóm tiếp tục mở rộng phân tích lên toàn bộ trình tự ADN của bộ gen vi khuẩn. Trong cụm gen kháng quen thuộc, một đoạn mã hóa protein chưa rõ chức năng đã hiện ra – một trình tự chưa từng xuất hiện trong bất kỳ kho dữ liệu di truyền quốc tế nào về kháng kháng sinh. Không hồ sơ. Không tiền lệ. Nhưng sự có mặt của nó là thật và đầy nghi vấn.
Đoạn gen ấy được nhóm nghiên cứu đặt tên là ILR-VIN [2] – viết tắt của Imipenem-Linked Resistance – Vietnam. Để kiểm tra giả thiết này, nhóm đã biểu hiện ILR-VIN trong một dòng vi khuẩn không mang các gen kháng. Kết quả cho thấy dòng mang ILR-VIN có khả năng sống sót cao hơn đáng kể trong môi trường chứa Imipenem so với các dòng đối chứng. Mặc dù chưa thể xác định chính xác cơ chế tác động, dữ kiện này cho phép đặt ra giả thuyết rằng ILR-VIN có thể liên quan đến khả năng kháng kháng sinh chưa từng được mô tả.
Một điểm đáng chú ý khác là ILR-VIN nằm trên đoạn ADN chứa nhiều gen kháng khác – một đặc điểm thường gặp ở các plasmid có khả năng lan truyền giữa các chủng vi khuẩn. Dù vẫn cần thêm nghiên cứu để xác minh, phát hiện này đặt ra cảnh báo về nguy cơ lan rộng của một gen kháng chưa từng được nhận diện – một ẩn số di truyền có thể làm thay đổi bản đồ kháng thuốc hiện tại nếu không được giám sát kịp thời.
Hệ sinh thái công cụ sinh tin học – chuỗi phân tích hiện đại phục vụ giám sát kháng thuốc
Để không bỏ sót bất kỳ tín hiệu di truyền nào từ hàng trăm bộ gen vi khuẩn, nhóm nghiên cứu của Học viện Quân y đã xây dựng một hệ sinh thái công cụ sinh tin học mã nguồn mở, hoạt động như một chuỗi phân tích khép kín, mạnh mẽ nhưng linh hoạt, có thể ứng dụng cả trong nghiên cứu lẫn thực hành lâm sàng.
Hành trình xử lý dữ liệu bắt đầu từ Pasa – công cụ thông minh giúp lựa chọn những bộ gen đại diện tối ưu từ hàng trăm chủng vi khuẩn [3]. Nhờ đó, hệ thống tránh được sự dư thừa thông tin nhưng vẫn đảm bảo giữ lại đầy đủ tính đa dạng di truyền cần thiết cho phân tích sâu hơn.
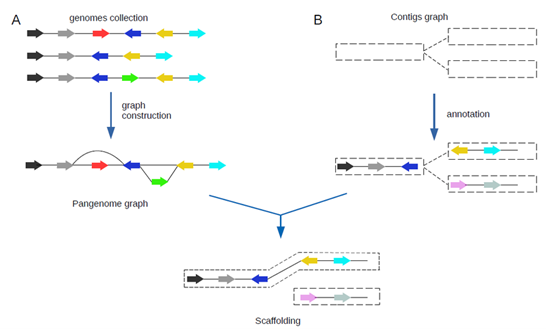
Từ tập đại diện này, PanTA – một mô hình học máy, sẽ tiếp nhận và xây dựng pangenome [4] một cách nhanh chóng và hiệu quả. Nhờ đó, các nhà khoa học có thể phân định rõ đâu là gen lõi tồn tại phổ biến, đâu là gen phụ hay biến thiên – những yếu tố thường gắn liền với khả năng kháng thuốc hoặc lan truyền.
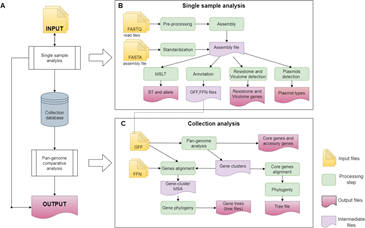
Tiếp theo, toàn bộ dữ liệu di truyền được chuyển sang AMRomics – một hệ thống phân tích toàn diện [5], tự động lắp ráp genome, định danh gen kháng, phân nhóm di truyền và so sánh đặc điểm giữa các chủng khác nhau. Đây là trục xử lý trung tâm của toàn bộ hệ thống.
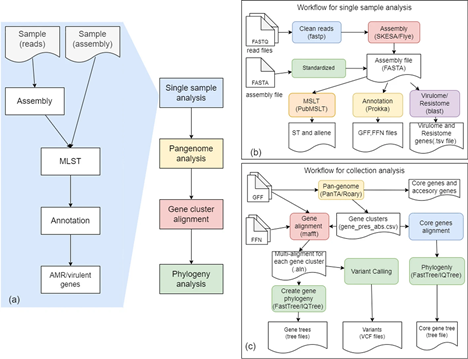
Để kết quả không chỉ dừng lại trong tay chuyên gia về sinh tin học, nhóm nghiên cứu phát triển thêm AMRViz – một giao diện trực quan [6], thân thiện, cho phép các bác sĩ lâm sàng, kỹ thuật viên hay sinh viên dễ dàng tra cứu, theo dõi và khai thác dữ liệu di truyền vi khuẩn một cách trực tiếp.
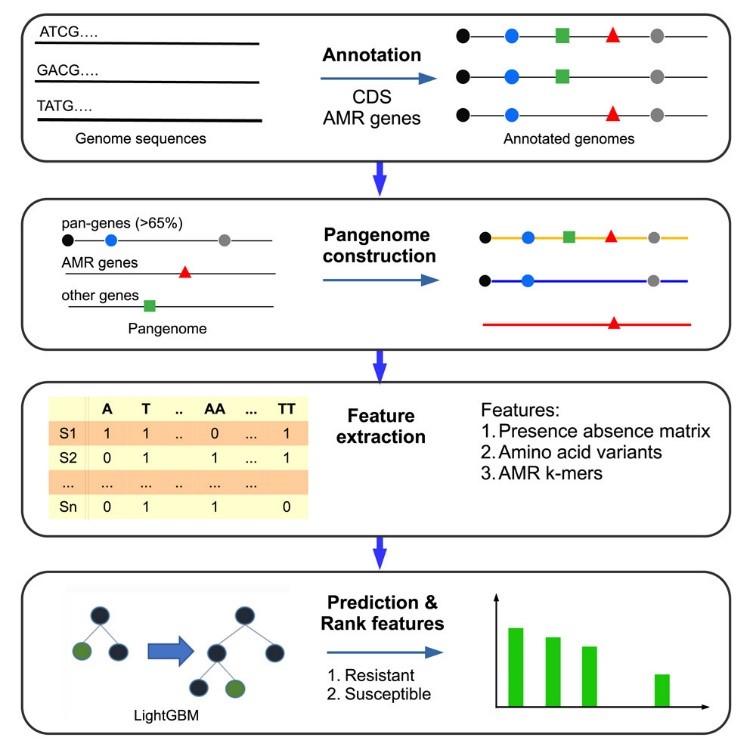
Bên cạnh đó, toàn bộ hệ dữ liệu pangenome được tích hợp vào PanKA, một mô hình trí tuệ nhân tạo tiên tiến [7] có khả năng tiên đoán kiểu hình kháng thuốc của vi khuẩn. Nhờ đó, bác sĩ hoàn toàn có thể dự báo mức độ đề kháng chỉ từ dữ liệu hệ gen, mở ra khả năng điều trị chính xác ngay cả trước khi có kết quả kháng sinh đồ.
Hệ sinh thái công cụ này không chỉ giúp tiết kiệm thời gian và chi phí phân tích mà còn có thể mở rộng ra các bệnh viện, trung tâm y tế dự phòng và hệ thống giám sát quốc gia. Các công cụ đã được công bố trên nhiều tạp chí khoa học quốc tế uy tín như Nucleic Acids Research, Genome Biology, iScience,… khẳng định khả năng làm chủ công nghệ sinh học tiên tiến của các nhà khoa học Việt Nam.
Kết luận
Từ việc giải trình và phân tích dữ liệu toàn bộ hệ gen vi khuẩn, nhóm nghiên cứu đã làm sáng tỏ những xu hướng kháng thuốc nguy hiểm, phát hiện gen kháng mới chưa từng được ghi nhận và xây dựng một hệ sinh thái công cụ hỗ trợ toàn diện cho việc giám sát và phòng chống.
Giải pháp này cho thấy khả năng Việt Nam hoàn toàn có thể chủ động tạo ra công nghệ của riêng mình để giải quyết một trong những vấn đề y tế nan giải nhất hiện nay. Dự án cũng là một hình mẫu cho hợp tác giữa nghiên cứu – công nghệ – chính sách trong bối cảnh toàn cầu hóa và dịch tễ học hiện đại.
Tác giả: TS.BS. Hồ Hữu Thọ, Học viện Quân y.
TS. Cao Minh Đức, Công ty CP Amromics.
Biên tập: Quỹ Đổi mới sáng tạo Vingroup (VinIF).
Tài liệu tham khảo
1. Le HTT, Hoang TT, Nguyen NAT, et al. Whole-Genome Sequencing Reveals Temporal Trends in Antibiotic Resistance Genes in Escherichia coli Causing Pediatric Urinary Tract Infections in Central Vietnam. Antibiotics. 2024; 13(9):830. https://doi.org/10.3390/antibiotics13090830.
2. Hoang, T.T., Le, H.T.T., Nguyen, S.N. et al. A novel gene linked to Imipenem resistance in E. coli isolate lacking known Imipenem-resistance genes. Scientific Reports 15, 9065 (2025). https://doi.org/10.1038/s41598-025-93587-0.
3. Van Hoan Do, Son Hoang Nguyen, Duc Quang Le et al. Pasa: leveraging population pangenome graph to scaffold prokaryote genome assemblies, Nucleic Acids Research, Volume 52, Issue 3, 9 February 2024, Page e15, https://doi.org/10.1093/nar/gkad1170.
4. Le, D.Q., Nguyen, T.A., Nguyen, S.H. et al. Efficient inference of large prokaryotic pangenomes with PanTA. Genome Biology, 25, 209 (2024). https://doi.org/10.1186/s13059-024-03362-z.
5. Le, D.Q., Nguyen, T.T., Nguyen, C.H. et al. AMRomics: a scalable workflow to analyze large microbial genome collections. BMC Genomics 25, 709 (2024). https://doi.org/10.1186/s12864-024-10620-8.
6. Le, D.Q., Nguyen, S.H., Nguyen, T.T. et al. AMRViz enables seamless genomics analysis and visualization of antimicrobial resistance. BMC Bioinformatics 25, 193 (2024). https://doi.org/10.1186/s12859-024-05792-9. 7. Do VH, Nguyen VS, Nguyen SH, et al. (2024). PanKA: Leveraging population pangenome to predict antibiotic resistance. iScience, 27, 110623. https://doi.org/10.1016/j.isci.2024.110623

