Di truyền ngoại gen (epigenetics) và methyl hóa DNA
Trong tế bào nhân thực, phân tử DNA được quấn quanh lõi histone (còn gọi là nucleosome) tạo thành sợi nhiễm sắc. Một nucleosome gồm các tiểu đơn vị histone H2A, H2B, H3 và H4. Một cách hiểu đơn giản, DNA được đóng gói trong cấu trúc nucleosome; các nucleosome có thể rất gần nhau làm cho sợi DNA co đặc lại hoặc chúng phân bố xa nhau làm cho sợi DNA tháo xoắn, bộc lộ các trình tự nucleotide ở dạng tự do (Hình 1). Việc quy định trạng thái co đặc hay tháo xoắn ở từng trình tự nucleotide, quy định sự tương tác giữa các trình tự nằm xa nhau trên một nhiễm sắc thể hay giữa các nhiễm sắc thể, gọi chung là quy định cấu trúc không gian của sợi nhiễm sắc, được kiểm soát bằng các cơ chế di truyền ngoại gen (epigenetics). Cấu trúc không gian của từng nhiễm sắc thể cũng như của toàn bộ hệ gen trong nhân được di truyền qua các thế hệ (phân bào giảm nhiễm), qua các lần phân chia tăng sinh (phân bào nguyên nhiễm). Khác với thông tin di truyền do trình tự nucleotide của phân tử DNA quyết định, di truyền ngoại gen tác động đến biểu hiện của gen ngay khi trình tự nucleotide trên phân tử ADN không hề thay đổi. Như vậy, thông tin di truyền gồm hai dạng: các mã di truyền bộ ba trên phân tử DNA và di truyền ngoại gen.

Trình tự nucleotide trên phân tử DNA gắn liền với thay đổi mức độ biểu hiện của tính trạng. Rất nhiều nghiên cứu đã chỉ rõ sai lệch một nucleotide, mất đoạn, thêm đoạn hay đảo đoạn trình tự nucleotide đều có thể là yếu tố quyết định tính trạng mới cũng như là nguyên nhân của nhiều căn bệnh hiểm nghèo [2]. Tuy nhiên, sai lệch của di truyền ngoại gen chủ yếu liên quan đến việc thêm/bớt những nhóm chức trên các nucleotide hoặc trên các acid amin của histone. Hiện nay, thêm/bớt nhóm CH3 vào cytosine đứng trong phức CpG ở động vật có vú đã được nghiên cứu nhiều nhất để đi đến kết luận methyl hóa DNA là một trong các cơ chế gây bất hoạt phiên mã của các gen. Thay đổi mức độ methyl hóa DNA trên một trình tự nucleotide tương ứng với sự co đặc lại hoặc tháo xoắn duỗi ra của sợi nhiễm sắc [1]. Một gen sẽ ở trạng thái sẵn sàng phiên mã khi trình tự nucleotide chứa gen đó được tháo xoắn. Ngược lại, gen sẽ bị bất hoạt khi bị quấn chặt quanh lõi histone [1]. Như vậy, biểu hiện của một gen không chỉ phụ thuộc trình tự nucleotide mà phụ thuộc vào cấu hình của đoạn nhiễm sắc chứa gen đó và các trình tự lân cận.
Methyl hóa DNA trong hệ gen được đặc biệt quan tâm đối với các trình tự giàu CpG (được gọi là đảo CpG-CpG insland) phân bố ở promoter các gen hoặc ở các trình tự DNA lặp như yếu tố DNA vận động, tâm động, vùng hạch nhân (Hình 2). Trong tế bào bình thường, mức độ methyl hóa DNA ở promoter các gen tiền ung thư (oncogenes) cao nhưng không có methyl hóa DNA ở promoter các gen ức chế ung thư (reppressor genes). Tương tự, các trình tự DNA vận động, vùng tâm động bị methyl hóa DNA cao tạo nên cấu trúc dị nhiễm sắc, nhờ đó đảm bảo được cấu trúc 3D và tính ổn định, trật tự của toàn bộ hệ gen. Tuy nhiên, trong tế bào già hóa, bệnh lý và ung thư, các hiện tượng này bị đảo ngược (Hình 2). Điều đặc biệt khác với đột biến nucleotide, tăng/giảm mức độ methyl hóa DNA có thể xảy ra thuận nghịch, động học của quá trình methyl hóa DNA biểu thị những diễn biến sớm nhất về thay đổi hoạt động của từng gen cũng như của toàn bộ hệ gen và chịu tác động của môi trường, chế độ dinh dưỡng và sinh hoạt [3]. Hiện nay, định lượng mức độ methyl hóa của một số gen đích và của một vài yếu tố DNA vận động đã trở thành dấu ấn hỗ trợ sàng lọc sớm, chẩn đoán, tiên lượng và cảnh báo tái phát của một số bệnh liên quan đến tuổi tác và ung thư.
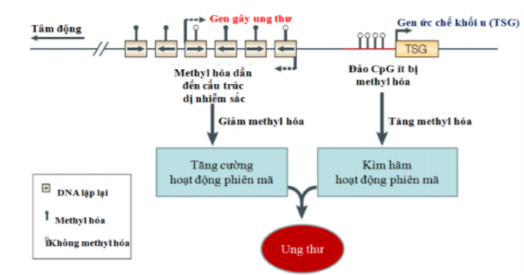
Dấu ấn methyl hóa DNA trong hỗ trợ chẩn đoán ung thư
Dấu ấn sinh học có giá trị trong sàng lọc, phát hiện sớm ung thư, dự báo chiều hướng diễn biến của bệnh ở mức độ phân tử. Dựa vào dấu ấn, bác sĩ lựa chọn phác đồ điều trị phù hợp (phẫu thuật, xạ trị, hóa trị), tiên lượng đáp ứng thuốc và khả năng thuyên giảm bệnh. Dấu ấn sinh học sử dụng trong chẩn đoán được xếp vào 3 nhóm lớn: (1) dấu ấn protein, (2) dấu ấn RNA, và (3) dấu ấn DNA gồm trình tự nucleotide và methyl hóa DNA. Trong đó, dấu ấn DNA có nhiều ưu điểm như thao tác đơn giản, không đòi hỏi lượng mẫu nhiều, DNA có thể tách từ các loại mẫu khác nhau (mẫu dịch, mẫu rắn, mẫu đúc paraffine) và có độ nhạy cao khi áp dụng kỹ thuật khuếch đại PCR. Đặc biệt, xu thế hiện nay phát triển các dấu ấn sử dụng cho mẫu sinh phẩm không hoặc ít xâm lấn (máu ngoại vi, nước tiểu, dịch, mẫu phân) để có thể thu nhận mẫu đơn giản cho sàng lọc định kỳ, giảm thiểu rủi ro đối với người có nhu cầu khám sức khỏe.
Methyl hóa xảy ra ở các phức CpG gắn liền với sự bất hoạt các gen ức chế khối u (tumor suppressor genes) được tìm thấy trong nhiều loại ung thư khác nhau (ung thư vú, phổi, tử cung, tiền liệt tuyến, đại trực tràng,…). Những gen ức chế khối u bị methyl hóa, do đó không hoạt động, có thể là các gen tham gia sửa chữa DNA (gen BRCA1), gen khử độc tố gây ung thư (gen GSTP1) hoặc là gen tham gia kiểm soát chu trình tế bào (gen RASSF1A). Methyl hóa DNA có thể chỉ xảy ra ở một số gen trong một loại bệnh ung thư. Ngược lại, methyl hóa xảy ra ở một gen trong nhiều loại ung thư. Ví dụ như, methyl hóa promoter BRCA1 chỉ xuất hiện ở ung thư vú, ung thư buồng trứng mà không phát hiện được ở các loại ung thư khác. Điều này cho thấy tính đặc hiệu của methyl hóa BRCA1 đối với hai loại ung thư đặc thù của phụ nữ. Ngược lại, methyl hóa promoter RASSF1A xảy ra với tỷ lệ cao ở nhiều loại ung thư khác nhau và được xem là dấu hiệu xảy ra sớm trong các tế bào ác tính, trong khi GSTP1 bị methyl hóa chủ yếu ở ung thư tuyến tiền liệt (~90%) nhưng ít hơn ở các loại ung thư khác (ở ung thư vú < 40%) [4].
Cùng với hiện tượng methyl hóa quá mức DNA (hypermethylation) ở vùng promoter các gen, methyl hóa dưới mức (hypomethylation) ở các trình tự DNA lặp, các yếu tố DNA vận động cũng xảy ra trong các tế bào ung thư. Khi bị methyl hóa dưới mức, sự di chuyển xảy ra dễ dàng làm cho nhiễm sắc thể bị thay đổi cấu trúc, hệ gen mất tính ổn định và một số gen sẽ hoạt động bất bình thường. Những hiện tượng này được quan sát thấy trong rất nhiều loại ung thư. Ví dụ như, methyl hóa dưới mức xảy ra ở yếu tố vận động LINE-1 (long interspersed nuclear element-1) làm cho LINE-1 di chuyển gây mất ổn định hệ gen. Trong tế bào bình thường, LINE-1 bị methyl hóa nhưng trong tế bào ung thư, LINE-1 bị khử methyl. Mức độ methyl dưới mức của LINE-1 liên quan đến các giai đoạn phát triển của ung thư đại trực tràng, ung thư vú, ung thư gan, ung thư dạ dày và ung thư phổi. Đáng chú ý, LINE-1 là DNA lặp lại chiếm đến khoảng 17 % genome/tế bào, cho nên mức độ methyl hóa LINE-1 được xem là đại diện cho mức độ methyl hóa của toàn bộ hệ gen [4].
Ngoài ra, dấu chuẩn methyl hóa DNA còn cho phép tiên lượng khả năng sống của bệnh nhân ung thư [5]. Đặc biệt, kháng hóa trị hoặc xạ trị là vấn đề thường gặp trong quá trình điều trị bệnh nhân ung thư; điều này khiến phác đồ điều trị gặp nhiều khó khăn. Mối liên quan giữa mức độ methyl hóa DNA và tính kháng hóa trị, ví dụ như kháng lại platini, cisplatin đã được xác nhận trong điều trị ung thư phổi, ung thư đại trực tràng, ung thư vú [5]. Trong những năm gần đây, thuốc điều trị đích có tác dụng làm thay đổi methyl hóa DNA đang được phát triển mạnh và đưa vào thử nghiệm lâm sàng. Ví dụ như, thuốc hoạt hóa phản ứng khử nhóm methyl trên DNA đã được Cơ quan thuốc và thực phẩm Mỹ (US Food and Drug Administration – FDA) cho phép sử dụng với bệnh nhân ung thư máu, ung thư lympho tế bào T [6]. Xác định mức độ methyl hóa BRCA1 được chỉ định làm xét nghiệm cho bệnh nhân ung thư vú để lựa chọn phác đồ điều trị với thuốc ức chế PARP (PolyAdenosine Diphosphate-Ribose Polymerase). Như vậy, mức độ methyl hóa DNA là chỉ thị tham chiếu hỗ trợ cho tiên lượng, đưa ra phác đồ điều trị đích phù hợp với từng loại bệnh ung thư [7].
Dấu ấn methyl hóa DNA sử dụng mẫu sinh phẩm không xâm lấn
Mẫu sinh phẩm không xâm lấn, ví dụ như mẫu máu, dịch, nước tiểu, mẫu phân đều chứa DNA, được gọi là DNA tự do (cfDNA). cfDNA đều chứa các thông tin di truyền và dấu ấn methyl hóa DNA đặc hiệu của tế bào tiết ra cfDNA. Hiện nay, một số bộ sinh phẩm phân tích dấu chuẩn methyl hóa DNA đã được cơ quan thuốc và thực phẩm Mỹ (FDA) cho phép sử dụng để sàng lọc, chẩn đoán sớm ung thư, đặc biệt với những bệnh ung thư chẩn đoán sớm giúp bệnh nhân có cơ hội khỏi bệnh cao như ung thư đại trực tràng, tuyến tiền liệt, cổ tử cung,… Hầu hết các bộ sinh phẩm này đều sử dụng mẫu sinh phẩm không xâm lấn. Để sàng lọc sớm ung thư đại trực tràng, 2 bộ sinh phẩm dựa trên dấu chuẩn methyl hóa DNA đã được FDA cấp phép sử dụng. Bộ sinh phẩm EpiProcolon phân tích methyl hóa gen Septin 9 (SEPT9) sử dụng máu ngoại vi đã được thử nghiệm ở Mỹ, châu Âu và Trung Quốc. Kết quả từ 25 thử nghiệm trên 9927 bệnh nhân ung thư đại trực tràng cho thấy EpiProcolon có độ nhạy, độ đặc hiệu và giá trị AUC tương ứng với 0.71; 0.92 và 0.88 [8]. Ngoài ra, bộ sinh phẩm ColoGuard sử dụng mẫu phân để phân tích dấu chuẩn methyl hóa 2 gen NDRG4 và BMP3 được FDA cấp phép từ 2014, được Hiệp hội Ung thư Mỹ (American Cancer Society) và Tổ chức đặc nhiệm về dịch vụ phòng bệnh Mỹ (U.S Preventive Service Task Force) đưa vào hướng dẫn sử dụng do có độ nhạy và độ đặc hiệu tối ưu nhất trong số các dấu ấn phân tử chẩn đoán ung thư đại trực tràng. Thử nghiệm kit ColoGuard với 9989 bệnh nhân cho độ nhạy phát hiện ung thư đạt 92.3%, đặc biệt phát hiện giai đoạn tiến triển ung thư (advanced precancerous lesions) đạt 42.4% trong khi dấu chuẩn FIT chỉ đạt 73.8% và 23.8% [9]. Đặc biệt, tỷ lệ phát hiện polyp có mức độ tăng sản cao (high-grade dysplasia), polyp không cuống (serrated sessile polyps) đường kính 1 cm của bộ kit ColoGuard là 69.2% và 42.4 %, trong khi xét nghiệm FIT chỉ đạt 46.2% và 5.1 % [9]. Hơn nữa, được sự hỗ trợ từ kết quả ColoGuard, kết quả nội soi có giá trị chẩn đoán tốt hơn [10]. Tuy nhiên, bộ sinh phẩm này được chỉ định sử dụng ở các phòng xét nghiệm do chính hãng sản xuất lựa chọn mà chưa thương mại hóa rộng rãi.
Trong trường hợp ung thư cổ tử cung, hướng dẫn tầm soát loại ung thư này ở Mỹ và châu Âu đều khuyến cáo thực hiện xét nghiệm tế bào học (pap smear) và xét nghiệm HPV với thời gian định kỳ 6-12 tháng tùy thuộc kết quả dương tính của một hoặc cả 2 xét nghiệm [44, 45]. Tuy nhiên, dấu ấn methyl hóa DNA, sử dụng mẫu dịch phết âm đạo do phụ nữ tự lấy mẫu, xác định bởi bộ kit WID™-qCIN đã được đưa vào trong chương trình sàng lọc ung thư cổ tử cung ở một số quốc gia phát triển như Thụy Điển, Úc [11].
Bên cạnh các bộ sinh phẩm đã được phép thương mại, một số bộ sinh phẩm sử dụng dấu chuẩn methyl hóa DNA đang trong giai đoạn thử nghiệm lâm sàng. Ví dụ như kit PredictMDx sử dụng dấu chuẩn methyl hóa gen MGMT (mã hóa O6-Methylguanine-DNA methyltransferase) đã được NCCN (National Comprehensive Cancer Network) khuyến cáo sử dụng trong hướng dẫn điều trị ung thư não [12], hoặc kit Colvera sử dụng dấu chuẩn methyl hóa BCAT1 (mã hóa enzyme Branched Chain Amino Acid Transaminase 1) và IKZF1 (mã hóa protein điều hòa có cấu trúc Zing finger) để hỗ trợ chẩn đoán sớm sự tái phát của ung thư đại trực tràng [13].
Phân tích tỷ lệ methyl hóa DNA bằng kỹ thuật định lượng đặc hiệu methyl qMSP (quantitative methylation specific PCR)
Kĩ thuật Real-time MSP (qMSP) được sử dụng phổ biến để định lượng tỷ lệ methyl hóa DNA (mDNA), là sự kết hợp giữa phương pháp xử lí bisulfite và thiết kế mồi đặc hiệu cho cytosine bị methyl hóa cho phản ứng realtime PCR. Đầu tiên, DNA được xử lý với bisulfite có tác dụng biến đổi cytosine không bị methyl hóa thành uracil, trong khi cytosine bị methyl được giữ nguyên (Hình 3). Sau xử lý, chỉ có các sợi đơn DNA mà tất cả cytosine không nằm trong phức CpG đều biến đổi thành uracil; DNA bị methyl hóa có CpG còn DNA không bị methyl hóa thì CpG biến đổi thành UpG (UpG sẽ chuyển thành TpG sau phản ứng PCR). Bằng cách này, DNA bị methyl hóa được phân biệt với DNA không bị methyl hóa nhờ các mồi đặc hiệu được thiết kế dựa vào trình tự giàu CpG [14].
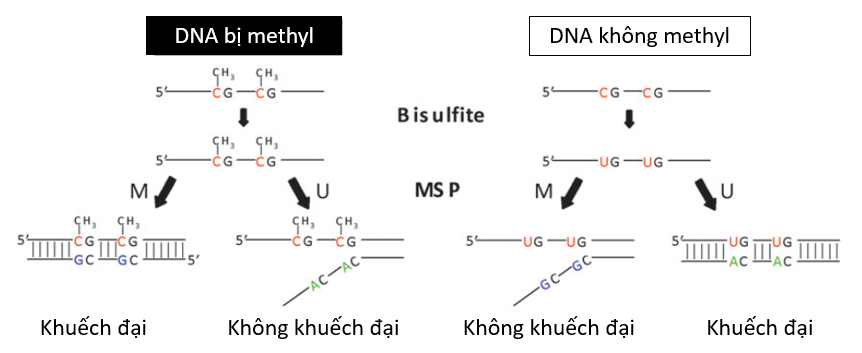
Tỷ lệ mDNA được xác định bằng phương pháp định lượng tương đối, bản chất là so sánh số lượng trình tự đích bị methyl hóa với tổng số trình tự đích [15, 16]. Như vậy phương pháp định lượng tương đối cần phải có mẫu chuẩn với tỷ lệ methyl hóa đã biết. Mẫu chuẩn có chứa trình tự đích là trình tự bị methyl hóa và trình tự tham chiếu đại diện cho tổng số DNA sau xử lí bisulfite, cả hai trình tự đều được biết rõ nồng độ hoặc số lượng bản sao [15]. Mẫu chuẩn có thể được tạo bằng cách phối trộn các plasmid mang đoạn chèn trình tự đích và trình tự tham chiếu hoặc sử dụng các mẫu chuẩn thương mại. Hiện nay, mô hình toán học được sử dụng phổ biến nhất trong định lượng tương đối là công thức Livak, 2-∆∆Ct [15]. Công thức này so sánh tỉ lệ giá trị Ct của trình tự tham chiếu với trình tự đích trong mẫu chuẩn và trong mẫu nghiên cứu, giá trị này nhân với tỉ lệ methyl hóa của mẫu chuẩn (đã biết trước do chủ động phối trộn) sẽ được kết quả cuối là tỉ lệ methyl hóa trong mẫu nghiên cứu [16].
Hầu hết các bộ kit phân tích dấu chuẩn mDNA cho chẩn đoán, tiên lượng ung thư đều xây dựng dựa vào kỹ thuật qMSP do có độ chính xác, độ nhạy cao và thiết bị rất phổ biến, dễ sử dụng ở các phòng xét nghiệm.
Nghiên cứu methyl hóa DNA ở Việt Nam
Những công bố đầu tiên ở Việt Nam về nghiên cứu mDNA đều liên quan đến tìm kiếm các chỉ thị phân biệt ung thư với bệnh không ung thư hoặc tình trạng khỏe mạnh. Ví dụ như nghiên cứu của Trịnh Hồng Thái & cs. (2013), Truong Kim Phượng & cs. (2014) tập trung phân tích methyl hóa gen APC và BRCA1 liên quan đến ung thư vú và ung thư đại trực tràng [17, 18]. Năm 2022, tác giả Nguyễn Hữu Thịnh & cs. đã sử dụng hệ thống array làm giàu trình tự chứa CpG bị methyl hóa và giải trình tự NGS cho phép phân tích methyl hóa cfDNA ở ung thư đại trực tràng giúp tăng tỷ lệ phát hiện sớm loại ung thư này ở bệnh nhân Việt Nam [19]. Công bố của tác giả Võ Thi Thương Lan & cs. (2022) và Nguyễn Phương Anh & cs. (2024) đã định lượng tỷ lệ methyl hóa gen SHOX2 ở cfDNA làm chỉ thị nhận biết ung thư phổi [20, 21]. Những kết quả đầu tiên này góp phần làm sáng tỏ tính khả thi của việc sử dụng dấu chuẩn methyl hóa DNA để chẩn đoán ung thư; từ đó thúc đẩy các nghiên cứu ứng dụng dấu chuẩn này vào xét nghiệm cận lâm sàng và hỗ trợ mô bệnh học để chẩn đoán sớm, tiên lượng chính xác và điều trị đích đúng bệnh ung thư.
Tài liệu tham khảo
- Rosa S, et al. (2013). Biology, 2: 1378-10. doi:10.3390/biology2041378
- Tycko B. (2002). Ann NY AcadSci, 983: 43-54. doi: 10.1111/j.1749-6632.2003.tb05961.x
- Liu J, et al. (2023). Environ Geochem Health, 45: 7543–68. doi:10.1007/s10653-023-01749-8
- Robertson K. (2005). Nat Rev Genet, 6:597-10. doi: 10.1038/nrg1655
- Walker R, et al. (2023). Adv Cancer Res, 158: 41–71. doi: 10.1016/bs.acr.2022.12.001
- Kaminskas E, et al. (2005). Oncologist, 10:176–82. doi: 10.1634/theoncologist.10-3-176.
- Zhu J, et al. (2024). Cancer Biol Med, 21: 111–116. doi: 10.20892/j.issn.2095-3941.2023.0403
- Nian J, et al. (2017). Clin Transl Gastroenterol, 8, e216; doi:10.1038/ctg.2016.66
- Imperiale TF, et al. (2014). N Engl J Med, 370:1287-97. doi: 10.1056/NEJMoa 1311194.
- Johnson DH, et al. (2017). Gastrointestinal Endoscopy, 85: 657-665.e1 doi: 10.1016/j.gie.2016.11.012
- Schreiberhuber L, et al. (2024). Nat Med. Doi: 10.1038/s41591-024-03014-6.
- Gilbert MR, et al. (2014). N Engl J Med, 370:699-708
doi: 10.1056/NEJMoa1308573.
- Murray DH, et al. (2017). J Appl Lab Med, 2:165-5. doi: 10.1373/jalm.2017.023135.
- Herman JG, et al. (1996). Proc Natl Acad Sci USA. 93:9821-9826. doi: 10.1073/pnas.93.18.9821
- Livak KJ, et al. (2001). Methods. 25:402-408. doi: 10.1006/meth.2001.1262.
- Thomassin H, et al. (2004). Nucleic Acids Res. 32:e168. doi: 10.1093/nar/gnh166
- Truong PK. et al. (2014). Asian Pac J Cancer Prev. 15:9607-9610. doi: 10.7314/apjcp.2014.15.22.9607.
- Dimberg J, et al. (2013). Oncol Lett. 5:25-30. doi: 10.3892/ol.2012.967.
- Nguyen HT, et al. (2022). Future Oncol. 18:3895-3912. doi: 10.2217/fon-2022-1041.
- Vo TTL, et al. (2022). Mol Biol Rep. 49:3413-3421. doi: 10.1007/s11033-022-07172-z.
- Phuong NA, et al. (2024). Biomolecules. 14:729. doi: 10.3390/biom14060729.

