Một trong những trụ cột không thể thiếu để xây dựng một nền Y học chính xác là bộ gen tham chiếu. Trải qua một lịch sử hơn 13 năm được tổng kết qua bài viết Bộ gen tham chiếu đã được xây dựng như thế nào?, quá trình hoàn thiện bộ gen tham chiếu của loài người đã đạt một số thành tựu nhất định. Với sự tiến bộ không ngừng nghỉ của khoa học – công nghệ, bộ gen tham chiếu liên tục được cập nhật theo hướng ngày càng chính xác hơn, tổng hợp được nhiều thông tin hơn để biểu diễn khái quát nhất cho bộ gen của loài người. Nhưng cũng vì quá trình cập nhật này, đã sinh ra không ít các phiên bản khác nhau bộ gen tham chiếu. Điều này có thể không ảnh hưởng đến một số phân tích hoặc có thể gây ra kết quả phân tích sai khác lớn trong lĩnh vực tin sinh học. Bài viết này sẽ góp phần mở ra cái nhìn tổng quan về bộ gen tham chiếu và sự khác nhau giữa các phiên bản thông dụng.
“Hình dáng” của gen tham chiếu
Gen tham chiếu (tiếng Anh: Reference Genome hoặc Reference Assembly) là một cơ sở dữ liệu dạng số của chuỗi axit nucleic, được lắp ráp (assembly) bởi các nhà khoa học, xem như một tập hợp gen của một cá thể sinh vật lý tưởng cho một loài. Vì được lắp ráp từ chuỗi DNA của một nhóm cá thể tham gia thí nghiệm, gen tham chiếu không hoàn toàn biểu diễn gen của một cá thể nào. Thay vào đó, gen tham chiếu cung cấp một thể khảm đơn bội của các chuỗi DNA khác nhau từ mỗi người tham gia thí nghiệm.
Định dạng đơn giản nhất của một bộ gen tham chiếu là một tệp fasta chứa các chuỗi axit nucleic hoặc axit amin, được chia làm nhiều “đoạn liên tiếp” (contigs, thường là các Nhiễm Sắc thể). Tuy nhiên, thông tin này chỉ đủ cho một loại phân tích là bắt cặp trình tự (alignment). Các loại tệp khác – tệp chú giải gen (genome annotation) GTF hoặc GFF cho phép nhiều loại phân tích xuôi dòng (downstream analysis) hơn vì chúng thể hiện được các thuộc tính của gen như toạ độ vùng phiên mã, exon, intron,…
Một số phiên bản bộ gen tham chiếu
Hiện nay, các phân tích, bài báo đã xuất bản trong lĩnh vực sinh học nghiên cứu liên quan đến gen người hoặc trong lĩnh vực tin sinh học phần lớn sử dụng hai phiên bản chính là hg19 (lấy tên khác là GRCh37 – Genome Reference Consortium Human Build 37, đặt tên theo lần họp thứ 37 của hội nghị này) hoặc hg38 (GRCh38). Tuy nhiên, với mỗi phiên bản, lại có rất nhiều phụ bản, được phát hành bởi các đơn vị nghiên cứu khác nhau.
Các phụ bản của hg19
GRCh37 (NCBI)
GRCh37, tên đầy đủ là Genome Reference Consortium Human Build 37, được đặt theo tên lần họp thứ 37 của hội nghị Genome Reference Consortium. Bản chính thức được xây dụng bởi Genome Reference Consortium, công bố vào ngày 27/02/2009. Tệp chứa bộ gen tham chiếu chính thức được phát hành và quản lý bởi NCBI (National Center for Biotechnology Information).
Trong phụ bản này, tên các nhiễm sắc thể số 1 tới 22 được đánh tên NC_00000a.b với a tương ứng từ 1 tới 22 và b là số định danh của phiên bản. Tương tự nhiễm sắc thể X, Y lần lượt có a=23, a=24.
Hiện tại, phụ bản này được NCBI cập nhật tới lần 13, tên tệp tin là GCF_000001405.25_GRCh37.p13_genomic.fna.gz với MD5sum 46e212080d30b1a24abec3eab36dbacd.
Nguồn chính thức:
- Các lần cập nhật ở đường dẫn này.
- Tệp chứa bộ gen tham chiếu FASTA
- Thư mục chứa các tệp liên quan GCF_000001405.25_GRCh37.p13
b37
Viện nghiên cứu Broad tạo ra một bộ gen tham chiếu mới dựa trên GRCh37 của NCBI, được đặt tên là b37. So với bản gốc, b37 có một vài thay đổi như tên các đoạn liên tiếp, và những bases có độ tin cậy thấp được chuyển thành kí tự N theo mã IUPAC.
Trong phụ bản này, tên các nhiễm sắc thể số 1 tới 22 được đánh tên tương ứng từ 1 tới 22. Nhiễm sắc thể X, Y lần lượt là X, Y.
Nguồn chính thức:
humanG1Kv37
Đây là phụ bản được sử dụng trong quá trình phân tích của 1000 Genomes Project. Phụ bản humanG1Kv37 tương đương với phiên bản b37 nhưng không chứa các đoạn mồi (decoy) cho gammaherpesvirus 4 ở người (NC_007605).
Trong phụ bản này, tên các nhiễm sắc thể số 1 tới 22 được đánh tên tương ứng từ 1 tới 22. Nhiễm sắc thể X, Y lần lượt là X, Y.
Nguồn chính thức:
- Tệp chứa bộ gen tham chiếu FASTA
- Thư mục chứa các tệp liên quan phase2_reference_assembly_sequence
hg19 (UCSC)
Trường đại học California ở Santa Cruz (UCSC) tạo ra một phụ bản hg19 dựa trên GRCh37. Phụ bản có tệp gen tham chiếu là hg19.fa.gz với MD5sum: 806c02398f5ac5da8ffd6da2d1d5d1a9.
Trong phụ bản này, tên các nhiễm sắc thể số 1 tới 22 được đánh tên tương ứng từ chr1 tới chr22. Nhiễm sắc thể X, Y lần lượt là chrX, chrY.
Nguồn chính thức:
- Tệp chứa bộ gen tham chiếu FASTA
- Thư mục chứa các tệp liên quan Golden Path
Các phụ bản của hg38
GRCh38 (NCBI)
GRCh38, tên đầy đủ là Genome Reference Consortium Human Build 38, được đặt theo tên lần họp thứ 38 của hội nghị Genome Reference Consortium. Bản chính thức được xây dụng bởi Genome Reference Consortium, công bố vào ngày 28/02/2019. Tệp chứa bộ gen tham chiếu chính thức được phát hành và quản lý bởi NCBI (National Center for Biotechnology Information). Hiện tại, phiên bản này được NCBI cập nhật tới lần 13, tên tệp tin là GCA_000001405.28_GRCh38.p13_genomic.fna.gzvới MD5sum: f28b7146e0f30efa58447eceb32620a3.
Trong phụ bản này, tên các nhiễm sắc thể số 1 tới 22 được đánh tên CM000a.2 với a tương ứng từ 663 tới 684. Tương tự nhiễm sắc thể X, Y lần lượt có a=685, a=686.
Nguồn chính thức:
- Các lần cập nhật ở đường dẫn này. Tệp chứa bộ gen tham chiếu FASTA
- Thư mục chứa các tệp liên quan GCA_000001405.28_GRCh38.p13
GRCh38 Resource bundle (Broad Institute)
Phụ bản này ngoài hệ gen tham chiếu, còn bao gồm những cơ sở dữ liệu tiêu chuẩn để phân tích dữ liệu giải trình gen người thông qua GATK – Genome Analysis Toolkit. Hiện nay Viện nghiên cứu Broad đang góp phần đưa GRCh38/hg38 thành tiêu chuẩn phân tích gen trên thế giới thông qua bộ sở dữ liệu tiêu chuẩn này. Dự án The International Genome Sample Resource (IGSR) cũng sử dụng bộ dữ liệu này làm cơ sở để phân tích trong Giai đoạn 3.
Trong phụ bản này, tên các nhiễm sắc thể số 1 tới 22 được đánh tên tương ứng từ chr1 tới chr22. Nhiễm sắc thể X, Y lần lượt là chrX, chrY.
Nguồn chính thức:
So sánh chi tiết
Ngoài việc thay đổi lớn về tọa độ, điểm làm hg38 trở nên hữu ích khi phân tích so với hg19 là số lượng lớn các vùng thay thế (alternate regions). Các vùng thay thế này mang tên dạng “*_alt”, miêu tả những chuỗi trình tự tồn tại phổ biến trong quần thể loài người nhưng lại có khác biệt tương đối lớn với trình tự trên hg38. Việc xuất hiện những vùng thay thế này đã giúp việc phân tích các quần thể khác nhau trên thế giới trở nên chính xác hơn.
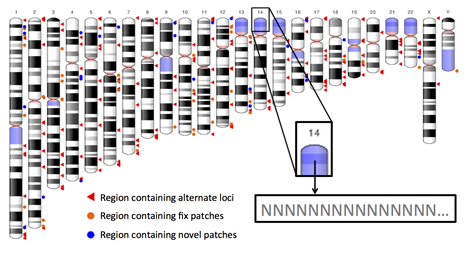
Ngoài ra, còn có sự khác biệt ở tên của các đoạn liên tiếp, biểu diễn vùng tâm động (centromere) và gen ti thể (mitochondrial genome).
Ở trong bài viết này, chúng tôi trực tiếp so sánh các đoạn liên tiếp giữa các phiên bản bộ gen tham chiếu thông qua tính giá trị tổng kiểm của mã hóa md5 (md5checksum). Trước tiên, chúng tôi đã tiến hành chuẩn hóa cách biểu diễn các trình tự trong các phụ bản và phiên bản khác nhau:
- Chuyển toàn bộ chữ in thường thành in hoa (chữ in thường thể hiện vùng repetitive)
- Chuyển toàn bộ kí tự khác A, T, G, C, N thành N. Lý do vì một số phiên bản có thể sử dụng kí hiệu của IUPAC.
Kết quả được miêu tả trong tệp sau:
Từ kết quả trên, chúng tôi rút ra một số quan sát:
- Phụ bản humanG1Kv37 có thêm đoạn mồi tổng hợp có tên “hs37d5” so với phụ bản b37 của Broad Institute.
- Cả 2 phiên bản GRCh37 và GRCh38 của NCBI đều không chứa chrEBV hay NC_007605 (gammaherpesvirus 4 ở người) so với các phụ bản khác. Ngoài ra, trong những “đoạn liên tiếp chính” (primary contigs), các phiên bản do NCBI phát hành này cũng khác hầu hết các phụ bản ở nhiễm sắc thể Y.
- Phụ bản hg19 của UCSC khác các phụ bản khác của hg19 ở gen ti thể M, trong khi lại giống nhiễm sắc thể Y với GRCh37 của NCBI.
Ngoài ra trong phiên bản GRCh38, phụ bản của NCBI khác với Broad Institute ở nhiễm sắc thể số 5, số 14, số 19, số 21, số 22. Phân tich cụ thể cho thấy các khác biệt này chủ yếu nằm ở kí hiệu của chuỗi trên vùng tâm động.
Bình luận mở rộng
Một kết quả khá bất ngờ cho thấy mặc dù cùng phiên bản, nhưng mỗi phụ bản phát hành bởi một đơn vị nghiên cứu khác nhau lại không hoàn toàn trùng khớp. Điều này đặc biệt ảnh hưởng đến khả năng tái lập kết quả giữa các nghiên cứu khác nhau, nhất là với những biến dị hiếm gặp trên những vùng đặc biệt.
Thêm vào đó, mặc dù phiên bản mới hơn của bộ gen tham chiếu là GRCh38 đã được cập nhật từ năm 2013, nhưng cho đến nay vẫn rất nhiều phân tích sử dụng phiên bản cũ hơn là GRCh37 (phát hành năm 2009). Nguyên nhân chủ yếu của việc này do khá nhiều công cụ, bộ cơ sở dữ liệu vẫn chưa cập nhật, nên các phân tích vẫn phải sử dụng bộ gen tham chiếu cũ. Các phân tích sử dụng phiên bản khác nhau có sự khác biệt đáng kể về toạ độ, cũng như độ tin cậy của phân tích, điều này cũng gây ra không ít khó khăn cho việc thống nhất, chia sẻ kết quả. Điều này, bên cạnh lý do về công nghệ, cũng là một phần lý do khiến GRC vẫn chưa cập nhật phiên bản tiếp theo của hệ gen tham chiếu ở người.
Tại Việt Nam, từ năm 2018, khi bắt đầu những bước đầu tiên của dự án giải mã 1000 hệ gen người Việt, Viện Nghiên cứu Dữ liệu lớn VinBigdata qua nghiên cứu kĩ lưỡng, đã tin tưởng và sử dụng bộ gen tham chiếu GRCh38 được đóng gói trong Resource bundle của Broad Institute. Qua đó, dự án có thể tận dụng được những bộ cơ sở dữ liệu và công cụ mới nhất, đồng thời đảm bảo được mức độ tin cậy cao với dữ liệu đầu ra.
Nguồn tham khảo:
GRCh37 hg19 b37 humanG1Kv37 – Human Reference Discrepancies
Human genome reference builds – GRCh38 or hg38 – b37 – hg19
Tác giả: Anh-Vu Mai-Nguyen
Người hướng dẫn: Thanh Nguyen

